热图 troubleshooting
热图问题,会持续更新
热图算是一个比较多的需求,基础使用上手比较快,但是想要美化的满意,需要下一番功夫。 这里使用 pheatmap 来画图
library(pheatmap)
library(AnnoProbe)
ls("package:AnnoProbe") # 查看包自带数据和函数
ex<-GSE95166$genes_expr
ex<-GSE95166$genes_expr
pheatmap(ex)
group_list=c(rep("N",4),rep("P",4))
ac<-data.frame(group_list)
n<-t(scale(t(ex[1:200,])))
n[n>2]=2
n[n<-2]=2
tiff("heatmap_200.tiff", width = 100, height = 100, units = 'in', compression ="lzw",res = 300)
# tiff("heatmap_200.tiff", paper="a4", units = 'in', compression ="lzw",res = 300)
# pdf("Your-file2.pdf", paper="a4", width=1000, height=1000)
#保存图片
pheatmap(n)
dev.off()
#修改细节
bk = unique(c(seq(-2,2, length=100)))
mycol<-colorRampPalette(c("white","tomato"))(100)
# mycol = colorRampPalette(c("navy", "white", "firebrick3"))(100)
```{r}
注意看以下的表演 生成注释所用数据框;
```{r}
ano_col<-data.frame(Cluster =factor(paste0('Cluster',cutree(p1$tree_col,3))))
#cutree切割出来的返回值十分像百分位定义饭后的值得结构,我这里的数据只有8个样本
order<-p1$tree_col$order # 获取聚类之后的顺序;
col_labels<-p1$tree_col$labels # 获取原始样本名
row.names(ano_col)=col_labels
ano_row<-data.frame(gene =factor(paste0('Cluster',cutree(p1$tree_row,9))))
order_row <- p1$tree_row$order
row_labels <- p1$tree_row$labels
row.names(ano_row)=row_labels
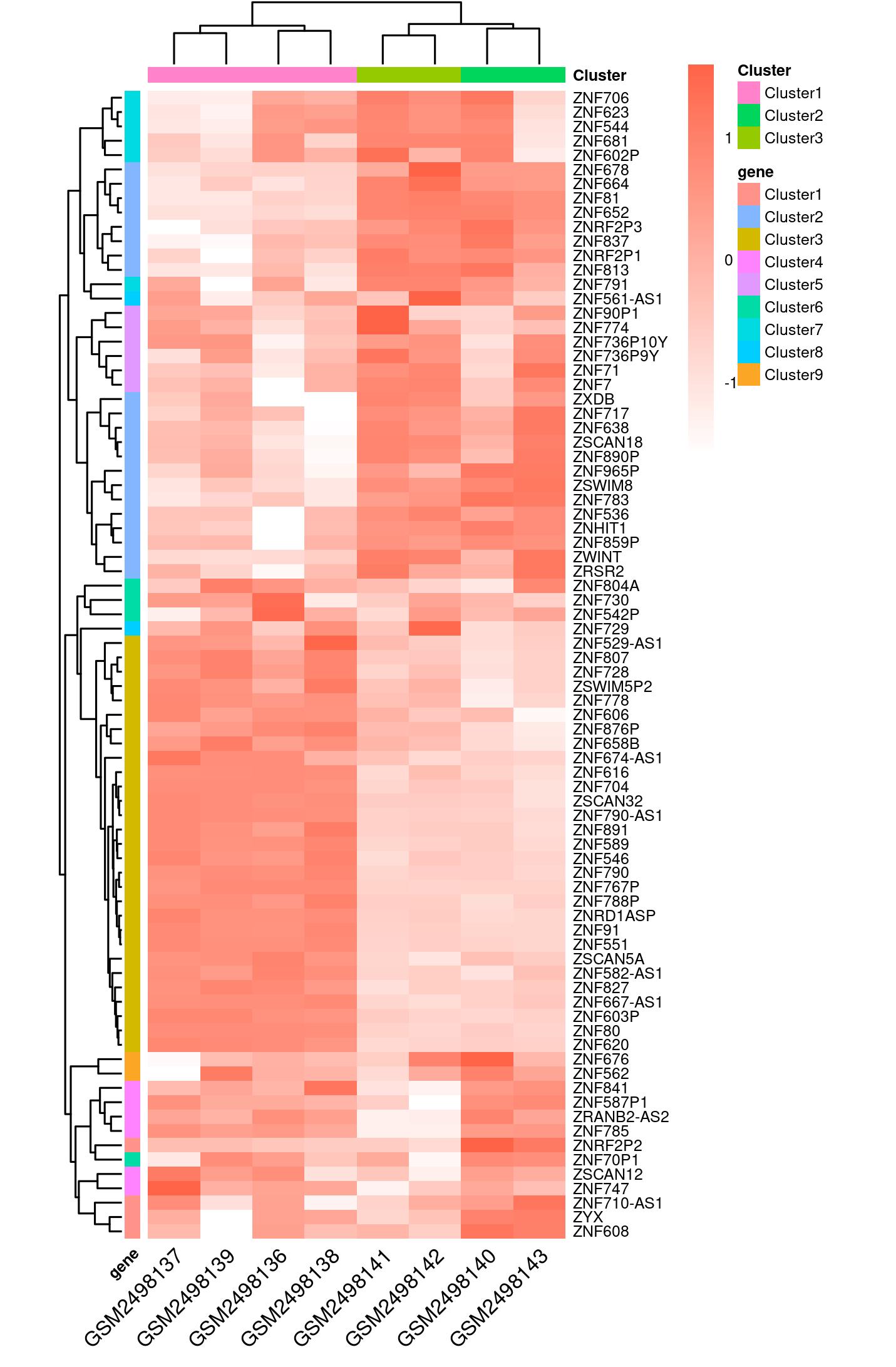
p1=pheatmap(n)
p2<-pheatmap(n,
cluster_rows=T, cluster_cols=T,
scale = "column",# 是否标准化,行、列或者none
border_color ="NA",#设置格子边框颜色,或者 NA 不显示
color = mycol,#使用的颜色风格
show_rownames=T,
legend_breaks=c(-2.5,-1.25,0,1.25,2.5), #设置注释条的break点的位置,即标签的颜色范围
legend_labels=c("-2.5","-1","0","1","2.5"),#设置注释的标签 名称,
breaks = bk,#定义颜色范围,使得图像区分更明显,
treeheight_row=20,#行 聚类树枝的高度
treeheight_col=20,# 列 聚类树枝的高度
fontsize=6, #字体大小
fontsize_col=8,#列名称字体大小
angle_col= 45,#列名称倾斜度,可以改的
annotation_col=ano_col,
#列注释是个数据框,如样本的分类,类型,聚类等等,可以有多列注释信息
annotation_row=ano_row,#行注释,同上;
annotation_legend=T,#是否显示旁边的注释信息
cellwidth = 20, cellheight = 5.5, # 格子大小,对美化很有用
filename = "top30heatmap.jpg",paper="a4" # 定义画布大小,很重要
)
# 保存数据
result<-n[order_row,order_col]
# 按照热图的顺序,导出作图数据;
result = data.frame(rownames(result),result)
# 整理表格,避免第一行数据错位;
colnames(result)[1] = "gene_id"
#导出数据到工作目录中;
write.table(result,file="Top30headmap.txt",
row.names=F,quote = F,sep='\t')